Le 9 octobre dernier, l’Académie royale des sciences de Suède annonçait l’attribution du prix Nobel de chimie 2024. Le professeur David Baker, reconnu pour ses travaux en « conception computationnelle de protéines », et l’équipe formée de Demis Hassabis et John M. Jumper, pour leurs avancées dans la « prédiction de la structure des protéines », se sont partagé cette prestigieuse distinction.
Cet article – Courant d’idées – est rédigé par Raphaël Gervais Lavoie, chargé de cours au Département de biochimie, chimie, physique et science forensique, et auxiliaire de recherche à l’Institut de recherche sur l’hydrogène de l’Université du Québec à Trois-Rivières.

Raphaël Gervais Lavoie est chargé de cours au Département de biochimie, chimie, physique et science forensique.
Professeur de biochimie à l’Université de Washington, David Baker et son équipe ont mis au point l’algorithme Rosetta, qui permet de prédire la structure des protéines de manière numérique. Les protéines sont des macromolécules constituées de séquences d’acides aminés. La prédiction de leur structure consiste à déterminer comment ces séquences s’agencent afin de former des structures tridimensionnelles. La prédiction de la structure des protéines constitue une partie fondamentale de la biologie computationnelle et revêt une importance cruciale dans plusieurs domaines (biologie, médecine, etc.) puisqu’elle facilite le développement de nouvelles enzymes et de médicaments innovants.
Parmi les méthodes contemporaines de prédiction, l’approche ab initio se distingue en permettant la conception de nouvelles structures fondées sur des principes physiques fondamentaux, sans se référer aux structures de protéines déjà connues. Cependant, la puissance de calcul requise pour réaliser des prédictions de manière ab initio est considérable. Pour y arriver, le groupe du professeur Baker a lancé le projet de calcul distribué Rosetta@Home. Ce projet consiste à segmenter des calculs de structures complexes en plusieurs parties, qui sont ensuite distribuées à un grand nombre d’ordinateurs. Ces ordinateurs, mis à disposition par des volontaires, exécutent les tâches requises et renvoient les résultats pour résoudre le problème initial. À ce jour, plus d’un million de collaborateurs participent à cet effort de recherche au travers du projet Rosetta@Home.
Le calcul volontaire distribué a été développé et popularisé au début des années 2000 grâce à l’infrastructure ouverte de Berkeley pour l’informatique en réseau (noté BOINC pour Berkeley Open Infrastructure for Network Computing). Celle-ci est utilisée pour de nombreux projets de recherche dans divers domaines scientifiques. Cette plateforme informatique permet aux chercheurs d’avoir accès à une importante puissance de calcul et contribue ainsi à l’avancement de la recherche et à l’accélération des découvertes.
Et qu’en est-il de l’apport de l’UQTR ?
Depuis plus de dix ans lors de la mise à niveau des systèmes informatiques, des ordinateurs de l’Université du Québec à Trois-Rivières (UQTR) sont recyclés pour constituer une grappe de calcul numérique. Cette infrastructure est d’abord utilisée par des étudiants du Département de biochimie, chimie, physique et science forensique pour, entre autres, modéliser les interactions des gaz dans des matériaux. Elle sert aussi à initier les étudiants aux outils de calcul parallèle ainsi qu’au calcul haute performance. Lorsque la grappe n’est pas mobilisée pour des projets académiques, sa puissance de calcul est mise à contribution dans divers projets de calcul distribué, notamment dans la recherche sur les ondes gravitationnelles, le cancer infantile, les énergies renouvelables et… les structures des protéines.
En effet, l’algorithme du professeur David Baker est aussi exécuté sur les ordinateurs de cette grappe de calcul, située sur le campus de l’UQTR. Grâce à cette contribution pour la recherche, l’UQTR se positionne parmi les 25 collaborateurs les plus actifs du projet Rosetta@Home. De plus, sa participation à l’ensemble des projets scientifiques de calcul distribué la place au 9e rang canadien, la positionnant largement en tête des universités au pays.
Ainsi, l’UQTR a contribué, à sa manière, au prix Nobel de chimie 2024 !
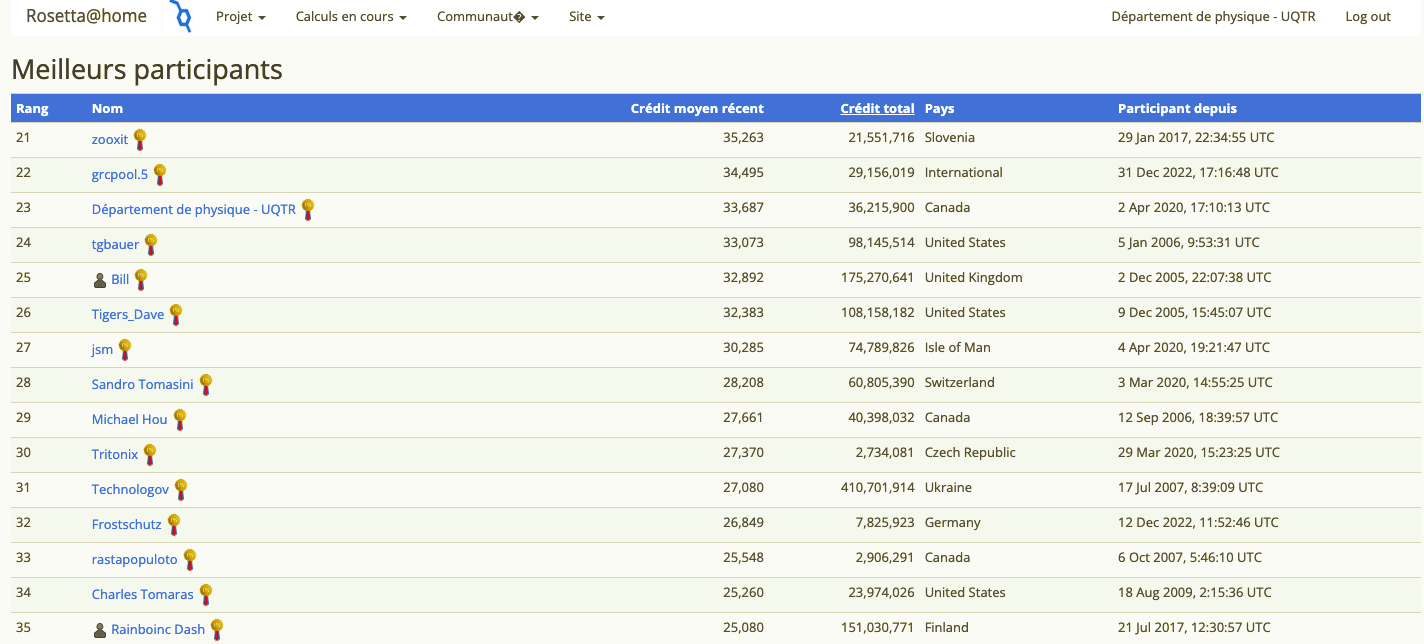
Classement des contributeurs les plus actifs au projet Rosetta@Home.
Sources et lectures complémentaires
https://www.nobelprize.org/prizes/chemistry/2024/press-release/
https://boinc.berkeley.edu/
https://boinc.bakerlab.org/rosetta/
https://www.boincstats.com/stats/-1/team/detail/165445/projectList
https://www.nature.com/articles/s41467-021-22276-z
https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1007507
https://link.springer.com/article/10.1007/s10723-019-09497-9
-
Courant d’idées permet à la communauté scientifique de l’UQTR de s’exprimer sur différents sujets et enjeux à travers une série d’articles vulgarisés pour le grand public. Consultez notre guide de rédaction.
Ouvert aux chercheurs, étudiants de cycles supérieurs et chargés de cours.


